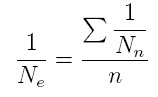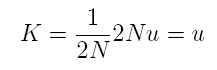Als Ergaenzung zu Argent23's Post "
Seeigelsex" auf
Holiday Junction poste ich hier das original Protokoll der Giglio-Exkursion von 2006. Es soll ja Leute geben, die ein Mikroskop ihr eigen nennen und unter dieser einen Vorraussetzungen ist das Ganze eine wirklich spannende Urlaubsbeschaeftigung. Vielleicht stolpert ja auch der eine oder andere Kursteilnehmer bei der Vorbereitung der Versuche ueber diese Posts... Viel Spass!
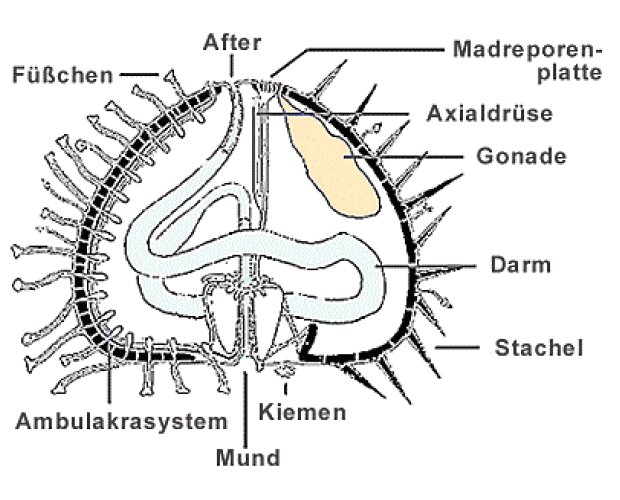
Larvalentwicklung der Echinodermata1. EinleitungStachelhäuter sind getrenntgeschlechtlich und pflanzen sich fort, indem sie Samenzellen und Eier direkt ins Wasser freisetzen. Das Geschlechterverhältnis ist dadurch bei vielen Arten zugunsten der Männchen auf etwa Drei zu Eins verschoben. Die meisten Arten haben pelagische Larven, die sich von Plankton ernähren. Im Gegensatz zu ihren Eltern sind die Larven bilateralsymmetrisch. Erst wenn sie sich auf dem Boden niederlassen verändert sich ihr Körper und zeigt die typische Radiärsymmetrie. In der Entwicklung der Larven wird außerdem der Urmund zum späteren After, und ein sekundärer Mund bildet sich, was die Echinodermata als Deuterostomier ausweist.
Seeigelembryonen werden daher, und weil sie leicht zugänglich und durchsichtig sind als Modellsystem für Entwicklungsvorgänge benutzt.
2.Gewinnung der GametenWir benutzten für unseren Versuch den Schwarzen Seeigel, Arbacia lixula, der im Mittelmeerraum und so auch auf Giglio an allen Felsküsten häufig vorkommt.
Da die Tiere sehr empfindlich auf Kontakt mit der Luft reagieren, wurde einmal sofort am Strand versucht Eizellen und Sperma zu gewinnen, ein zweites Mal wurden andere Tiere erst im Labor der Prozedur unterzogen. Die Tiere wurden mit der Oralseite nach oben in ein Becherglas, gefüllt mit sterilfiltriertem Meerwasser, gesetzt. Zum Sterilfiltrieren hatten wir das Meerwasser mit einer Spritze und einem geeigneten Aufsatz durch einen Filter mit einer Maschenweite von 0,2μm gepresst.
Den Seeigeln wurde nun etwa 2ml Kaliumchlorid mit einer Injektionsspritze durch die Mundöffnung in die Leibeshöhle injiziert, was eine Depolarisation und ein Ausstoßen der Gameten bewirken sollte.
Dazu verwendeten wir am Strand versehentlich 30%iges (circa 5M) im Labor später 0,5 molares KCl. Trotzdem kam es bei beiden Ansätzen zu einem Ausstoßen von Gameten, wobei nicht alle Tiere wie gewünscht reagierten. Es zeigte sich dass eine schräge Injektion tief ins innere des Tieres die größte
Erfolgswahrscheinlichkeit mit sich bringt. Dies könnte daher rühren, dass so das Kaliumchlorid direkt in die Gonade seitlich unter der Apikalseite gespritzt wurde.
Folgende Tabellen zeigen die Anzahl männlicher und weiblicher Tiere, die Keimzellen ausstießen. Die Zahl in der Spalte BG gibt an wie viele Tiere weder Spermien noch Eizellen abgaben.
Strand
BG---männlich---weiblich
6------------3---------------2
Labor
BG---männlich---weiblich
4------------4---------------2
Durch die hohe Anzahl von Tieren unbestimmten Geschlechts macht eine Statistik der Geschlechterverteilung keinen Sinn. Die Gameten wurden mit einer Pipette vom Boden der Bechergläser entnommen und getrennt, gekühlt aufbewahrt.
Entwicklung der EmbryonenWir gaben nun in zwei Petrischalen die am Strand erhaltenen Spermien zu den dort gewonnenen Eizellen und stellten eine der beiden Schalen kühl.
Ebenso verfuhren wir mit den erst im Labor erhaltenen Geschlechtszellen.
Nach der Befruchtung der bereits polar gebauten Eizelle(Polkörperchen am animalen Pol) wurde ein Eindringen weiterer Spermien durch eine Veränderung der Plasmamembran zur Befruchtungsmembran, die nun so genannte Corticalgranula als äußere Schutzschicht enthält, verhindert. Diese Befruchtungsmembran war bei allen Versuchsansätzen als dünner Kranz um die Zygote zu erkennen.
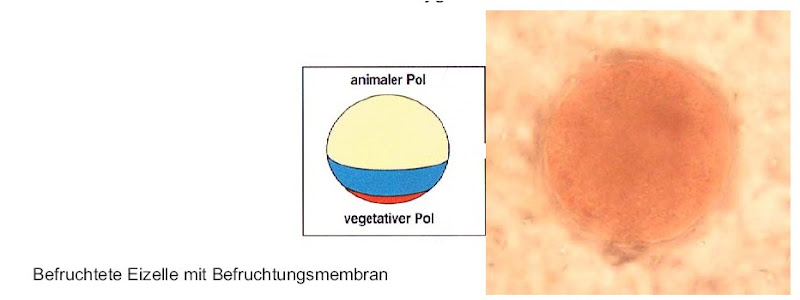
Auch die ersten beiden Furchungen, die das Ei entlang der animal-vegetativen Achse teilen konnten wir bei den bei Zimmertemperatur gelagerten Ansätzen nach ca.20 und 40 Minuten erkennen. Die im Kühlschrank gelagerten Embryonen erreichten nach etwa 20 Stunden dieses Stadium.

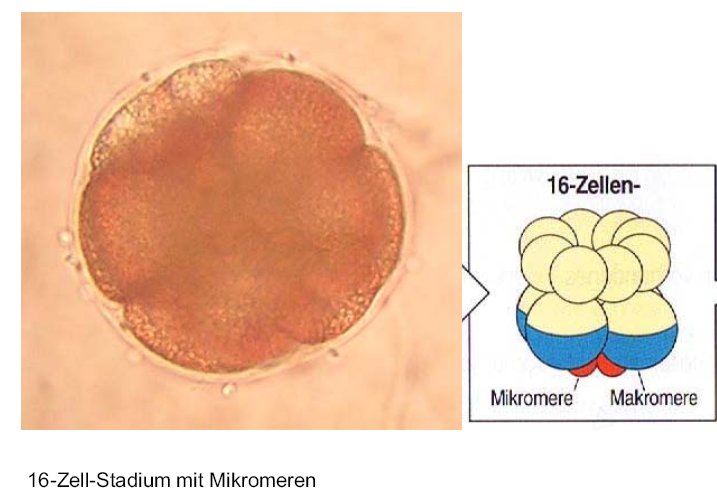
Nach etwa zwei Stunden hatten die Zimmertemperatur-Ansätze das 16-Zell-Stadium erreicht. Dieses entsteht nach der vierten inäqual verlaufenden Furchung und zeigt am vegetativen Pol vier kleine Mikromeren.
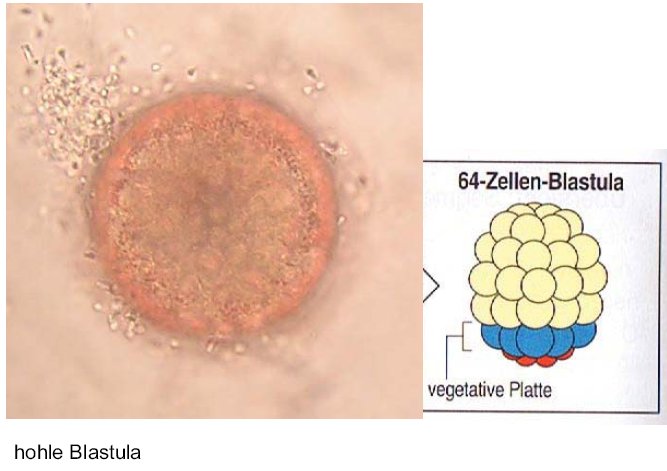
Nach etwa 5 Stunden befanden sich in den beiden bei Zimmertemperatur gelagerten Ansätzen bereits Blastulae. Auch die gekühlten Ansätze erreichten dieses Stadium, allerdings erst nach fünf Tagen.
Die darauf folgenden Stadien, zwischen Blastula und Larve, entwickelten sich über Nacht und konnten daher leider nicht dokumentiert werden.
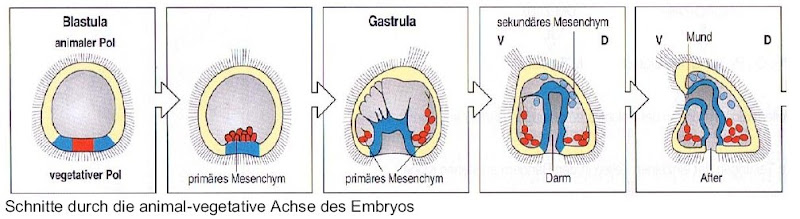
Bei Beginn der Gastrulation wanderten vom vegetativen Pol etwa 40 primäre
Mesenchymzellen in das innere der Blastula. Dort stülpte sich der Darm ein, der dann mit dem Mund, der sich von der gegenüberliegenden Seite eingestülpt hatte, verschmolz.
Nach etwa 48 Stunden konnten wir im Ansatz, mit den am Strand gewonnenen
Gameten, der bei Zimmertemperatur gelagert wurde, Pluteuslarven entdecken.
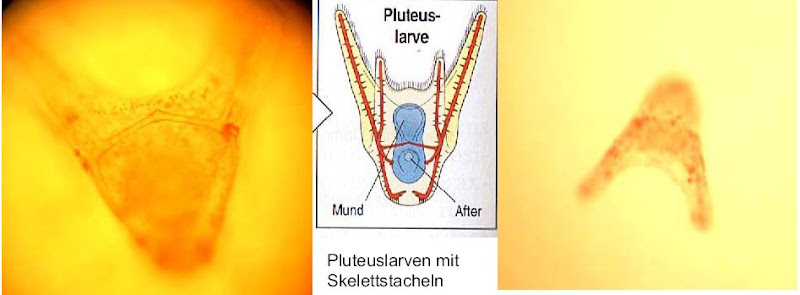
Die Embryonen des im Labor gewonnenen Ansatzes waren von Pilzsporen oder
anderen Verunreinigungen zerstört. Auch die beiden im Kühlschrank gelagerten Ansätze starben nach fünf Tagen im Blastulastadium ab.
Wahrscheinlich war nur beim ersten Sterilfiltrieren des Meerwassers unser Filteraufsatz richtig dicht.
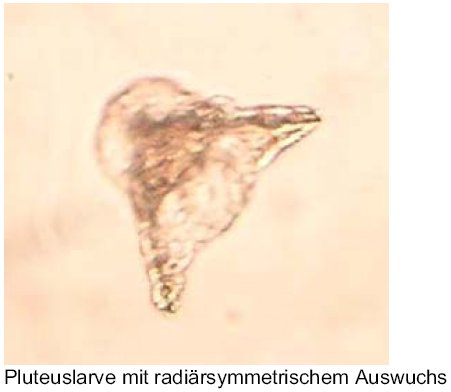
In unserem ersten Ansatz konnten wir nach drei Tagen sogar die beginnende
Metamorphose zum radiärsymmetrischen Tier beobachten. Es hatte sich am Boden der Petrischale mit einem radiärsymmetrischen Auswuchs angeheftet.