Während meiner Diplomarbeit habe ich neben meinem Interesse an Genomik auch einen Hang zu quantitativem Arbeiten entwickelt. Genauer gesagt sollte ich damals die Darmwand "meines [1]" Wurms elektronenmikroskopisch untersuchen und verschiedene Parameter dieses Epithels aus verschiedenen "experimentellen Gruppen" vergleichen(d vermessen). Ich habe mir daraufhin ein Programm beschafft mit dem ich die digitalisierten Bilder vermessen konnte und habe so eine riesigen Datensatz generiert.
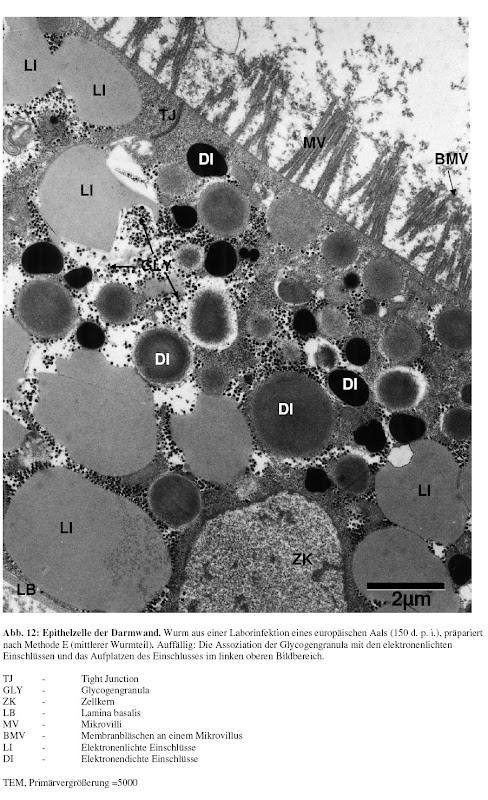
Die Analyse in SPSS gestaltete sich dann folgendermaßen:
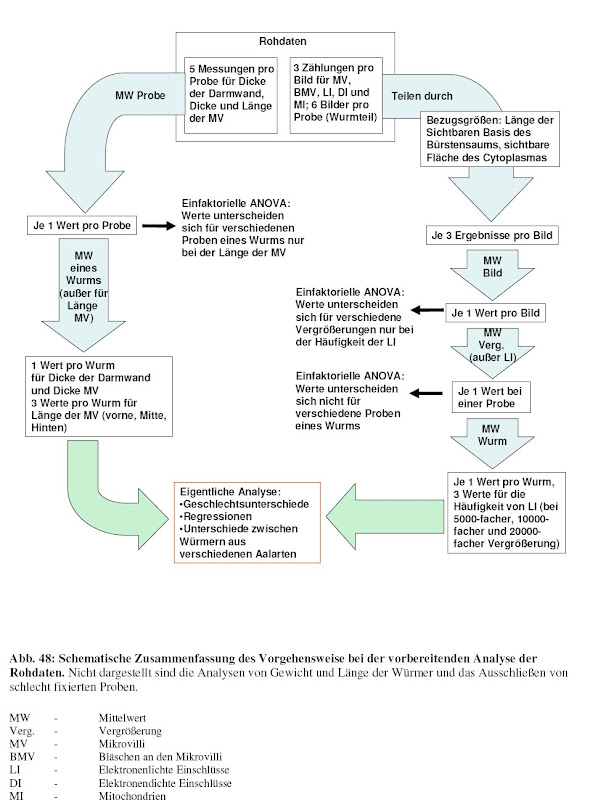
Alles was nach den erste beiden Pfeilen steht ist dabei was die Vorgehensweise bei der Datenauswertung betrifft bestenfalls nicht besonders elegant [2]. Effekte aller Variablen habe ich nacheinander in willkürlicher Weise auseinander "seziert".
Die korrekte Vorgehensweise wäre ein statistisches Modell auf die Daten anzupassen. Dabei würden dann schrittweise von einem Maximalmodell ausgehend nicht signifikante Variablen ausgeschlossen.
Einer der vielen Vorteile von R ist nun, dass es die Datenanalyse geradezu in Richtung einer solchen Modellierung lenkt.
Der Datensatz meiner Diplomarbeit ist für meine derzeitigen Fähigkeiten trotzdem noch etwas zu kompliziert. Ich beschäftige mich hauptsächlich mit Infektionsdaten, die ich Anfang des Jahres in Taiwan gesammelt habe. Es geht darum den Zusammenhang von Kapseln in der Darm- und Schwimmblasenwand mit der Infektion zu testen (die Kapseln sind tote Larven!).

Neben meiner statistischen Modellierung amplifizieren und sequenzieren wir ("mein" Diplomand Dominik) im Moment aus den Kapseln einige Gene für die wir sehr Nematoden-spezifische Primer haben.
[1] Sobald "mein" Transkriptom online ist lass ich die Anführungszeichen weg ;-)
[2] Ich wurde gerade dafür trotzdem von den Gutachtern meiner Arbeit gelobt. Das Hauptproblem an diesem Datensatz ist auch wirklich die pseudo-Replikation, darum die vielen Mittelwerte.

Keine Kommentare:
Kommentar veröffentlichen